Crear vacunas (y valorar su eficacia) desde un PC

Tendemos a clasificar la respuesta inmunitaria generada tras una vacunación como humoral (anticuerpos) o celular. Sin embargo, a pesar de que las respuestas de uno u otro tipo pueden llegar a ser predominantes, no son ni mucho menos excluyentes. En general, el rol más importante en la protección recae en la respuesta humoral, especialmente en el caso de las infecciones víricas. Así, la existencia de una cantidad suficiente de anticuerpos con capacidad neutralizante puede estar relacionada incluso con una inmunidad esterilizante -aquella que anula totalmente el virus e impide su replicación-. Entonces, en ausencia de anticuerpos, ¿el animal está totalmente desprotegido?
No todo es inmunidad humoral
Tomemos como ejemplo el virus de la gripe (influenza). Una característica muy importante de este virus es su elevada variabilidad genética; los antígenos -partes del virus que inducen una respuesta inmune- relacionados con la producción de anticuerpos pueden llegar a ser muy distintos entre dos virus influenza. Curiosamente, estas diferencias se han observado incluso entre virus de un mismo grupo.
Imaginemos dos virus: X e Y, de los cuales sabemos que poseen antígenos relacionados con la producción de anticuerpos muy diferentes entre sí. De forma simplificada, animales vacunados con X desarrollarán anticuerpos que probablemente no serán capaces de neutralizar a Y. A este fenómeno lo llamamos ausencia de anticuerpos de reacción cruzada. Así pues, a priori, cabría pensar que los animales vacunados con X no estarán protegidos frente a Y. Pero ¿y si no ocurriera así? ¿Y si los animales vacunados con X estuvieran protegidos frente al virus Y aun en ausencia de anticuerpos de reacción cruzada?
Existen estudios, tanto con virus de influenza humana como porcina, en los que se ha observado protección total o parcial -protección basada en la reducción de lesiones pulmonares y títulos virales- en ausencia de anticuerpos de reacción cruzada. En estos casos, parte de la protección podría atribuirse a las respuestas mediadas por los linfocitos T citotóxicos (respuesta celular). En nuestro ejemplo, las diferencias entre X e Y en los antígenos que inducen la producción de anticuerpos impide la existencia de anticuerpos de reacción cruzada. Sin embargo, si existiera similitud en aquellas partes del virus que determinan la respuesta celular, podría existir protección. Para poder evaluar a priori la similitud de los antígenos, y cómo se relacionarían con los receptores de las células del sistema inmunitario, podemos usar herramientas bioinformáticas.
La bioinformática y su aplicación en a la evaluación de la respuesta inmunitaria
De forma muy sencilla, definimos bioinformática como el área de la ciencia basada en la biología y la informática cuyo principal objetivo es gestionar, relacionar e interpretar un enorme volumen de datos procedentes de la genómica, la inmunología, etc.
Antes de la aplicación de la bioinformática en el tema que nos ocupa hoy, localizar epítopos -fragmentos específicos de los antígenos reconocidos por las células del sistema inmune, entre ellas los linfocitos T- suponía un proceso muy costoso desde todos los puntos de vista; había que sintetizar decenas, cuando no cientos, de segmentos de cada proteína del virus y posteriormente realizar los pertinentes análisis inmunológicos. Sin embargo, la bioinformática simplifica notoriamente este proceso, haciéndolo muchísimo más sencillo y barato; se realiza una predicción para escoger posibles candidatos y así en los análisis inmunológicos posteriores se evalúa solamente dichos candidatos. La combinación de bioinformática + análisis inmunológicos es pues un enfoque mucho más racional.
Aunque con la crisis sanitaria del SARS-COV-2 algunos de estos conceptos se han hecho más visibles, en realidad hace años que se aplican. De hecho, el primer estudio que aplicó herramientas bioinformáticas para la detección y posterior evaluación de epítopos de células T en la especie porcina data del 2009 (Díaz y colaboradores, 2009 ).
En un estudio más reciente (Gutiérrez y colaboradores, 2017 ), todos estos conceptos han sido aplicados mediante el algoritmo denominado EpiCC con el objeto de comparar epítopos T de diferentes cepas del virus de la influenza A en cerdos, y poder estimar la respuesta celular cruzada entre ellas. A nivel práctico, EpiCC puede ser útil para predecir sobre el soporte informático la eficacia de las vacunas frente a las cepas circulantes. Evidentemente, aparte de poder realizar predicciones, este tipo de herramienta aporta una información muy valiosa, la cual, junto a la que aportan los análisis filogenéticos y la reactividad cruzada de anticuerpos, puede facilitar la creación de vacunas más racionales y dirigidas.
En el análisis, en los que se comparan los epítopos compartidos entre cepas teniendo en cuenta también las secuencias de los receptores de las células del sistema inmune, EpiCC otorga una puntuación. Para marcar el umbral que debería poseer una vacuna (puntuación mínima) para definirla como protectora frente a una cepa concreta, se analizaron los resultados de seis estudios anteriores en los que los cerdos habían sido vacunados frente a influenza y posteriormente desafiados con una cepa de campo concreta. En cinco de estos estudios, la vacuna otorgaba protección -en términos de reducción de lesiones a nivel pulmonar y de títulos virales en pulmón e hisopo nasal- a pesar de inducir un nivel de anticuerpos de reacción cruzada muy bajo.
Gráfico 1. Definición del umbral para predecir la eficacia de la vacuna. La línea azul marca la puntuación obtenida mediante EpiCC entre la vacuna (H1γ FS) y cada cepa. Los umbrales de protección y protección parcial se definen en tres áreas: Blanco (protección), Gris claro (protección parcial) y Gris oscuro (sin protección). (P) indica las cepas frente a las cuales hubo protección y (PP) protección parcial, según los estudios experimentales. El resto de las cepas no han sido testadas in vivo. Gráfico extraído de Gutiérrez y colaboradores (2017).
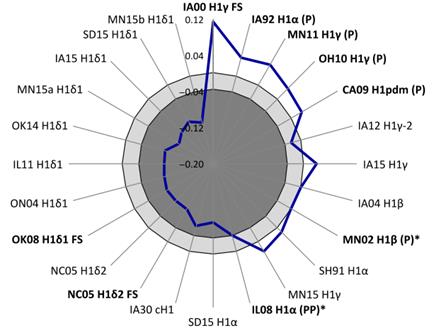
En el gráfico 1 se define el umbral (puntuación mínima) a partir del cual podríamos prever que la vacuna otorgaría protección o protección parcial. Como vemos, el gráfico predice, en función de las respuestas celulares, que la vacuna otorgaría protección frente a las cepas situadas en la zona blanca y parcial frente a las que están en la zona gris. En la tabla 1 se detallan las cepas usadas para marcar el umbral, así como su nivel de protección, tanto real (estudios experimentales) como estimada (puntuación EpiCC).
Tabla1. Estudios de desafío en animales vacunados frente a influenza (Tabla extraída de Gutiérrez y colaboradores (2017)).
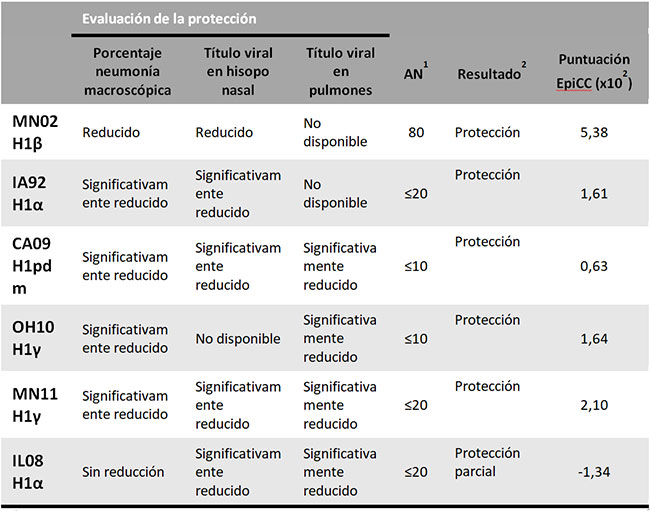
1 Anticuerpos neutralizantes frente a la cepa de desafío.
2 La vacuna se considera protectora si reduce la neumonía a nivel macroscópico así como el título viral en los hisopos nasales y/o en los pulmones recogidos en la necropsia. Si la vacuna reduce significativamente el título viral, pero no las lesiones pulmonares, se considera como parcialmente protectora.
Como puede observarse, existe una relación entre la predicción realizada mediante EpiCC y la protección real que otorgó la vacuna en los estudios experimentales, lo cual demostraría la utilidad de dicha herramienta.
Referencias:
Díaz I, et al. (2009). In silico prediction and ex vivo evaluation of potential T-cell epitopes in glycoproteins 4 and 5 and nucleocapsid protein of genotype-I (European) of porcine reproductive and respiratory syndrome virus. Vaccine. doi: 10.1016/j.vaccine.2009.07.029.
Gutiérrez AH, et al. (2017). T-cell epitope content comparison (EpiCC) of swine H1 influenza A virus hemagglutinin. Influenza Other Respir Viruses. doi: 10.1111/irv.12513.

